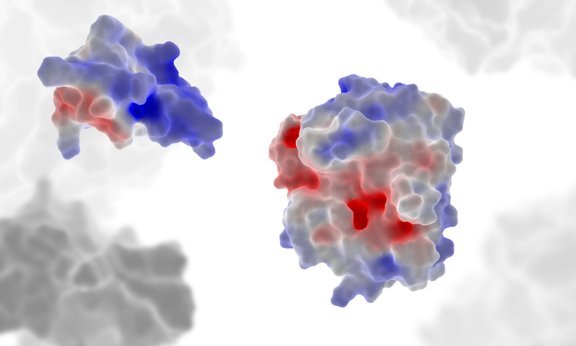
Enzyme gehören zu den wichtigsten Werkzeugen der belebten Natur. Die kleinen Biokatalysatoren beschleunigen diverse Umwandlungsprozesse und sind Teil jedes Organismus, jeder Zelle und dort beispielsweise in die Energiegewinnung des Körpers involviert. Auch sogenannte Proteasen gehören zu den Enzymen. Sie sind darauf spezialisiert, Proteine zu spalten. Im Verdauungssystem zerlegen sie alle Proteinanteile der ankommenden Nahrung und machen sie für die körperliche Verwertung bereit. Ganz ähnliche Proteasen sind auch an der Blutgerinnungsfunktion des Körpers beteiligt – als Teil der Gerinnungskaskade sind sie hochspezialisierte Elemente eines komplexen, selbstregulierenden Systems.
Verschiedene Arten von Proteasen können sich, obwohl in ihrer Form kaum Unterschiede bestehen, in ihrer Funktionalität stark unterscheiden. Manche sind sehr spezifisch und sprechen nur ein ganz bestimmtes Protein an, andere sind „promiskuitive“, nahezu universelle Schneidewerkzeuge, die so gut wie jedes Protein aufspalten, auf das sie treffen – eine Fähigkeit, die etwa in der Verdauung gefragt ist. Klaus R. Liedl, Leiter des Instituts für Allgemeine, Anorganische und Theoretische Chemie der Universität Innsbruck, und sein Team gehen diesen verschiedenen Eigenschaften der Proteasen auf den Grund. Die Forschenden nutzen dafür hoch entwickelte Computersimulationen, um herauszufinden, warum sich die Proteinwerkzeuge trotz ihrer nahezu identen Struktur so unterschiedlich verhalten.
Verbesserung von therapeutischen Antikörpern
Wenn man besser versteht und vorhersagen kann, welche Proteasen ganz spezifisch und welche universell funktionieren, lässt sich dieses Wissen in der Immunologie und Therapie anwenden, etwa bei Immunisierung und Therapie viraler Erkrankungen wie bei Infektionen mit HIV, Influenza oder Coronaviren, die sich durch Mutationen ständig verändern. „Die Proteasen sind für uns im Grunde nur ein Testsystem, das hilft, die physikalischen Vorgänge bei der Erkennung von Biomolekülen jeder Art besser zu verstehen“, erklärt der Wissenschaftler. „Beispielsweise könnte die Übertragung der Erkenntnisse auf die Proteinstrukturen von therapeutischen Antikörpern diese Biopharmazeutika in ihrer Wirkung deutlich zielgenauer machen.“ Wirkstoffe dieser Art sind Hoffnungsträger in vielen diagnostischen und therapeutischen Bereichen – allen voran im Kampf gegen Krebs.
Die Proteasen wurden als Gegenstand der Forschung gewählt, weil für sie aus vergangenen Wirkstoffforschungen eine besonders gute Datenlage vorhanden ist – inklusive Röntgenstrukturanalysen und ganzer Datenbanken zum Verhalten verschiedenster Varianten. Die Unterschiede zwischen spezifisch und promiskuitiv agierenden Proteasen liegen laut bisherigen Erkenntnissen vor allem in ihren dynamischen Eigenschaften begründet. Sie werden also erst offenbar, wenn die Biomoleküle in ihren Bewegungen erfasst werden können. Zu diesem Zweck kommen nun die Simulationswerkzeuge ins Spiel, die von Liedl und seinem Team entwickelt wurden und das Verhalten der Proteasen über eine möglichst lange Zeitspanne hinweg berechnen sollen.
Weit auseinanderliegende Zeitskalen überbrücken
„Die Kunst ist, den Proteasen möglichst lange zuzuschauen“, fasst Liedl zusammen. Doch genau das stellt die Forschenden vor eine große Herausforderung. „Das Problem dabei ist, dass die Zeitskalen der Simulation und des Phänomens, das wir uns ansehen wollen, sehr weit auseinanderliegen: Das simulierte Verhalten der beteiligten Atome läuft auf einer Femtosekunden-Zeitskala ab, also in Millionstel von Milliardstelsekunden. Die Phänomene, die uns interessieren, liegen aber im Millisekundenbereich, also ab einer Tausendstelsekunde.“ Zum Vergleich: Das ist so, als würde man das Verhalten eines Objekts, das sich im Sekundentakt bewegt, über zumindest 32.000 Jahre vorausberechnen wollen.
Eine der schwierigsten Aufgaben bei Forschungen in diesem Bereich ist also die Bereitstellung der notwendigen Rechenkraft, die eine solche Simulation in einer überschaubaren Zeit absolvieren kann. „Wir arbeiten mit einer Kombination aus lokaler Computerinfrastruktur, die wir selbst aufbauen und programmieren, und universitären Rechenzentren“, schildert Liedl. Die Forschungsgruppe erprobt zudem den Einsatz von Künstliche-Intelligenz-Systemen, um effizienter an die gewünschten Ergebnisse zu gelangen.
Verstehen die Forschenden besser, wie Dynamik und Struktur der Proteasen zusammenhängen, kommt man auch der Vision näher, die Erkennungscharakteristik von Biomolekülen gezielt zu designen. Im Fall der therapeutischen Antikörper könnte man diese gezielt anpassen, indem man die Genetik ihres Produktionssystems – bestimmte Zelllinien, die die gewünschten Proteine hervorbringen – verändert. Liedl: „In der Praxis würden wir also Mutationen bestimmter Gensequenzen vorschlagen, die die Eigenschaften der therapeutischen Antikörper in die gewünschte Richtung verändern und sie promiskuitiver, spezifischer oder aber auch haltbarer machen.“
Zur Person
Klaus R. Liedl ist Professor für Theoretische Chemie und Leiter des Instituts für Allgemeine, Anorganische und Theoretische Chemie der Universität Innsbruck. Der Wissenschaftler mit Studienabschlüssen in Chemie, Mathematik und Rechtswissenschaften lehrte zudem an Universitäten in Italien, Brasilien, Thailand und Indonesien. „Promiskuität und Spezifität von Proteasen“ ist Liedls siebentes FWF-Projekt, das mit 393.000 Euro gefördert wird und noch bis 2022 läuft.