Die Auswertung von Abwasserproben ist für die Beobachtung des epidemiologischen Geschehens, insbesondere was die Erkennung und Ausbreitung neuer Varianten anbelangt, hoch relevant: Was seit Pandemiebeginn vermutet wurde, konnten Wissenschaftler mehrerer österreichischer Forschungseinrichtungen nun mittels umfassender Daten belegen und hochrangig publizieren. Unter Federführung der Arbeitsgruppen um Andreas Bergthaler (CeMM und MedUni Wien), Heribert Insam (Universität Innsbruck) und Norbert Kreuzinger (TU Wien) nimmt Österreich international eine Vorreiterrolle im Bereich Abwasser-Monitoring ein. Die Universität Innsbruck ist an der Kollaboration von Anfang an, seit März 2020, maßgeblich beteiligt. „Damals hat die ganze Welt ja verzweifelt nach Reagenzien gesucht, es gab zu wenig Tests“, erzählt Nachwuchswissenschaftler Rudolf Markt vom Institut für Mikrobiologie der Uni Innsbruck. Beim Nachdenken, wie man die Anzahl der Proben verringern und gleichzeitig viel Menschen erreichen könnte, sei ihm die Idee gekommen, nach genomischem Virus-Material im Abwasser zu suchen. Der Nachweis von COVID im Abwasser gelang dem Doktoratsstudenten Markt aus der Forschungsgruppe von Andreas Wagner wenig später, zeitgleich mit weiteren österreichischen Kolleg*innen, worauf man sich zu einer beispiellosen, disziplinen- und institutionenübergreifenden Kooperation zusammentat. „Die Methode, die wir am Institut für Mikrobiologie 2020 zur Quantifizierung von Sars-CoV-2 erarbeitet haben, ist nach wie vor die Methode der Wahl in Österreich“, sagt Markt, dessen Expertise insbesondere im molekularbiologisch-methodischen Bereich der Aufkonzentrierung liegt. „Ich beschäftigte mich aber auch mit der Frage, wie wir die unterschiedlichen Virus-Mutationen im Abwasser finden und den jeweiligen Varianten zuweisen können“, schildert er eine große Herausforderung bei der Abwasseranalyse. Mittels Abwasserdaten lässt sich sogar die Verbreitung einzelner Varianten, der Varianten-spezifischen R-Wert, berechnen, wie die Erstautoren Amman und Markt im Fachjournal Nature Biotechnology erstmals zeigen können.
Patientenproben und Abwasser-Daten stimmen größtenteils überein
Mehr als 3.400 Abwasserproben aus über 90 kommunalen Einzugsgebieten bzw. Kläranlagen sequenzierten und analysierten die Wissenschaftler von Dezember 2020 bis Februar 2022 für die Studie, was einer wöchentlichen Abdeckung von mehr als 50 Prozent der österreichischen Bevölkerung entspricht. Mittels einer eigens entwickelten Software (Variant Quantification in Sewage designed for Robustness, kurz VaQuERo) konnten die Wissenschaftler die räumlich-zeitliche Häufigkeit von Virusvarianten aus komplexen Abwasserproben ableiten. Diese Ergebnisse wurden anschließend anhand epidemiologischer Aufzeichnungen von mehr als 311.000 Einzelfällen gemeinsam mit den Infektionsepidemiologen der AGES validiert. Erstautor Fabian Amman, Bioinformatiker in der Forschungsgruppe von Bergthaler am CeMM und der MedUni Wien, erklärt: „Unsere Ergebnisse bestätigen, dass trotz zahlreicher Herausforderungen bei der Abwasseranalyse die Ergebnisse einen sehr genauen Überblick über das Pandemiegeschehen eines ganzen Landes bieten. Für jede Woche und jedes Einzugsgebiet, in denen laut epidemiologischem Meldesystem eine bestimmte Variante zumindest einmal auftrat, sehen wir in 86% der Proben derselben Woche ein entsprechendes Signal im Abwasser. Umgekehrt sehen wir in rund 3% der Abwasserproben Varianten, die dem Patienten-basierten System entgangen sind.“
Die im Rahmen der Studie generierten Daten bieten eine Basis für die Vorhersage neu entstehender Varianten und machen den Reproduktionsvorteil bedenklicher Varianten besser kalkulierbar. Ein weiterer Vorteil des Abwassermonitorings ist zudem, dass auch asymptomatische Personen sowie Personen, die das Testangebot nicht nutzen, in den Daten erfasst werden.
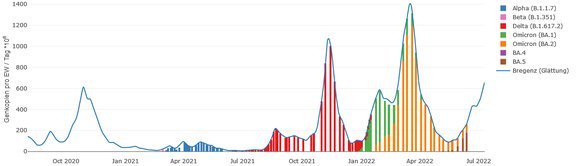
Die Grafik zeigt den Verlauf der Pandemie im Einzugsgebiet der Kläranlage Bregenz auf Basis von Abwasserdaten. Die blaue Linie entspricht der Anzahl an SARS-CoV-2 Fragmenten im Abwasser. Die bunten Balken entsprechen dem jeweiligen Anteil unterschiedlicher Varianten berechnet auf Basis der Resultate der Ganzgenom-Sequenzierung aus den Abwasserproben.
Erfolgsfaktor Zusammenarbeit
Durch die erfolgreiche Zusammenarbeit zwischen Forschungseinrichtungen und verschiedenen Behörden konnte in Österreich frühzeitig ein flächendeckendes Monitoring aufgesetzt werden. „Dies ist auch eine Erfolgsstory, was wissenschaftliche Zusammenarbeit schaffen kann. Im konkreten Fall war dies gekennzeichnet von früh begonnenen und erfolgreich weiter ausgebauten Kollaborationen zwischen CeMM und MedUni Wien, Universität Innsbruck, der Medizinischen Universität Innsbruck, der Technischen Universität Wien, der AGES sowie mehr als zehn weiteren Institutionen“, erklärt Forschungsgruppen-Leiter Andreas Bergthaler. Es sei zu erwarten, dass die Erkenntnisse über SARS-CoV-2 im Abwasser zukünftig auch auf die Analysen anderer Infektionserreger im Abwasser Anwendung finden werden.
Publikation: Wastewater surveillance of SARS-CoV-2 variants in Austria between December 2020 and February 2022. Fabian Amman, Rudolf Markt, Lukas Endler, Sebastian Hupfauf, Benedikt Agerer, Anna Schedl, Lukas Richter, Melanie Zechmeister, Martin Bicher, Georg Heiler, Petr Triska, Matthew Thornton, Thomas Penz, Martin Senekowitsch, Jan Laine, Zsofia Keszei, Peter Klimek, Fabiana Nägele, Markus Mayr, Beatrice Daleiden, Martin Steinlechner, Harald Niederstätter, Petra Heidinger, Wolfgang Rauch, Christoph Scheffknecht, Gunther Vogl, Günther Weichlinger, Andreas Otto Wagner, Katarzyna Slipko, Amandine Masseron, Elena Radu, Franz Allerberger, Niki Popper, Christoph Bock, Daniela Schmid, Herbert Oberacher, Norbert Kreuzinger, Heribert Insam, Andreas Bergthaler. Nature Biotechnology, 18 July 2022. DOI: 10.1038/s41587-022-01387-y