Fruchtfliege, Plattwurm und Zebrafisch helfen der Wissenschaft bei der Klärung vieler Fragen. Sie können mit einfachen Methoden gezüchtet und untersucht werden, haben kurze Entwicklungszyklen und lassen sich relativ einfach halten. Deshalb kommt diesen Modellorganismen in der biologischen und biomedizinischen Forschung eine große Bedeutung zu.
Biologen um Peter Ladurner vom Institut für Zoologie der Uni Innsbruck haben vor über zehn Jahren den Plattwurm Macrostomum lignano erstmals beschrieben. Entdeckt haben sie ihn, wie sein Name schon andeutet, in der oberen Adria. Hier lebt er in den Sandstränden und zeichnet sich durch hohe Stressresistenz aus. Er lebt im Salzwasser, kommt aber auch mit Brackwasser oder langer Trockenheit klar. Diese Robustheit macht ihn zum idealen Kandidaten für das Labor.
Ladurner hat mit seinem Team in den vergangenen Jahren zahlreiche Methoden entwickelt, um moderne Untersuchungen an dem Plattwurm durchführen zu können und ihn als Modellorganismus für Forschungen an Bioadhäsion, Stammzellen, Alternsforschung, Regeneration und Evolution zu etablieren. Gemeinsam mit Forschern aus Großbritannien und den USA gelang es Peter Ladurner 2015, das Genom des Plattwurms vollständig zu sequenzieren. „Die besondere Herausforderung lag dabei in der extrem hohen Anzahl an repetitiven Sequenzen im Genom. Erst durch die Verwendung modernster Sequenzierungsmethoden mit besonders langen Reads ist es uns gelungen, das Genom zu entschlüsseln“, erzählt der Zoologe. Eine verbesserte Version des Genom-Assemblies wird in der vorliegenden Arbeit vorgestellt.
Wichtiger Modellorganismus
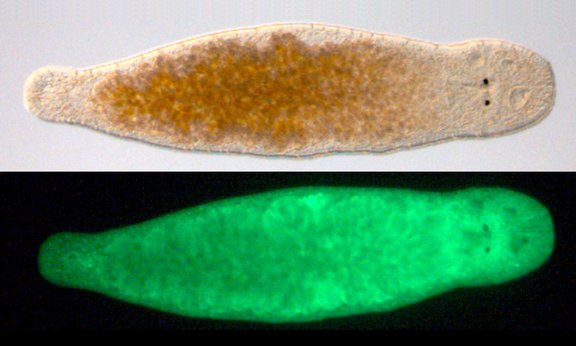
Schon früh hat Ladurner auch versucht, transgene Tiere zu züchten. Dabei wird künstliches Erbgut in Eizellen von Plattwürmern eingebracht, um bestimmte Proteine in der Zelle zum Beispiel mit Farbstoffen zu markieren. Die Forscher können dann entwicklungsbiologische Phänomene in den Tieren sehr einfach beobachten. „Sind die markierten Proteine im Organismus ausgeprägt, leuchten sie und können leicht identifiziert werden“, erklärt Ladurner. Diese Methode wurde nun weiterentwickelt und von Ladurner gemeinsam mit niederländischen Forschern in der Fachzeitschrift Nature Communications präsentiert. „Das ist ein wichtiger Schritt für die Entwicklungsbiologie“, freut sich Peter Ladurner. „Die Möglichkeit, transgene Tiere zu erzeugen, ist entscheidend, um eine Art als Modellsystem zu etablieren.“ Die Forscher zeigen in der aktuellen Arbeit zahlreiche unterschiedliche Regulationssequenzen, die bestimmte Proteine mit unterschiedlichen Farbstoffen markieren können. Um die neuen Modelle überprüfen zu können, hat Julia Wunderer aus dem Team von Ladurner mit einer komplementären Methode zahlreiche Vergleichsmuster erzeugt. Durch die Untersuchungen konnte außerdem das Genom noch genauer beschrieben werden.
Diese Arbeit könnte die Grundlage für eine breite Etablierung dieses ‚Innsbrucker Wurms’ als Modellorganismus in der biomedizinischen Forschung legen. Solche Organismen schaffen nicht nur die Basis für neue Erkenntnis in der Biologie, sondern liefern auch der medizinischen Forschung neue Möglichkeiten, die Funktion von bestimmten Genen zu identifizieren. Weitere Anwendung finden sie in der pharmazeutischen Industrie für die Produktion von Medikamenten und in der Biotechnologie.
Links
- Efficient transgenesis and annotated genome sequence of the regenerative flatworm model Macrostomum lignano. Jakub Wudarski, Daniil Simanov, Kirill Ustyantsev, Katrien de Mulder, Margriet Grelling, Magda Grudniewska, Frank Beltman, Lisa Glazenburg, Turan Demircan, Julia Wunderer, Weihong Qi, Dita B. Vizoso, Philipp M. Weissert, Daniel Olivieri, Stijn Mouton, Victor Guryev, Aziz Aboobaker, Lukas Scharer, Peter Ladurner, Eugene Berezikov. Nature Communications 2017
- Peter Ladurner
- Institut für Zoologie