Vor drei Jahren wurden vier neue Klassen von Ribozymen entdeckt. Ähnlich wie Enzyme beschleunigen diese RNA-Moleküle chemische Reaktionen in der Zelle. Bei der Beschreibung ihrer genauen Struktur und Funktion liefern sich Forschungsgruppen aus aller Welt derzeit einen Wettlauf. Ganz vorne dabei ist das Team um Ronald Micura am Institut für Organische Chemie. Bereits 2014 ist es ihnen gemeinsam mit Partnern in den USA gelungen, die Struktur des sogenannten Twister-Moleküls zu beschreiben. Dieses Ribozym ist weitverbreitet und kommt sowohl in Bakterien als auch in Eukaryoten vor. Es gewinnt für biotechnologische Anwendungen immer mehr an Bedeutung. Im vergangenen Jahr folgte dann die erstmalige Strukturbeschreibung des sogenannten Pistol-Ribozyms durch die Innsbrucker Forscher. Neben Twister und Pistol zählen noch Twister-Sister und Hatchet zu den neu entdeckten Klassen von Ribozymen. Deren Struktur und Funktionsweise waren bisher noch unbekannt.
In einer im Oktober im Fachmagazin Nature Communications erschienenen Arbeit hat die Forschungsgruppe um Ronald Micura gemeinsam mit Forschern in China nun die Struktur der Twister-Sister-Moleküle aufgeklärt. Das Sequenzdesign und die Synthesen für die Untersuchungen wurden in Innsbruck gemacht, die Kristallisation und Röntgenstrukturanalyse erfolgte durch die Wissenschaftler in China. „Die nun präsentierte Strukturbeschreibung von Twister-Sister liegt näher an der aktiven Form des Moleküls, als eine kurz zuvor veröffentliche Struktur einer anderen Gruppe. Es ist deshalb für Funktionsanalysen wesentlich interessanter“, sagt Ronald Micura. „Nun ist nur noch die Aufklärung der Funktionsweise des strukturell sehr flexiblen und damit schwer fassbaren Hatchet-Ribozyms offen.“ Auch an dessen Beschreibung arbeiten die Forscher bereits.
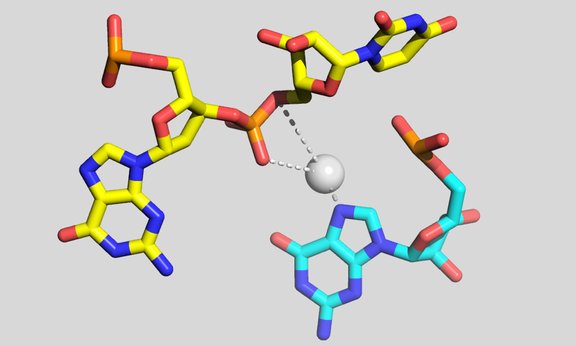
Die Gruppe um Micura hat nach der Strukturaufklärung 2016 auch das Pistol-Molekül weiter untersucht und dabei den chemischen Mechanismus aufgeklärt, der nun im Fachmagazin Angewandte Chemie veröffentlicht wurde. „Ribozyme schneiden sich selbst entzwei und aktivieren sich auf diese Weise“, erklärt Ronald Micura. „Wir konnten jene Nukleotide und ein Metallion im Pistol-Ribozym eindeutig identifizieren, welche für die chemische Reaktion der spezifischen Phosphorsäuredieesterspaltung entscheidend sind.“ Die funktionelle Aufklärung im Labor war sehr aufwändig, weil die Chemiker zahlreiche neue Varianten des Moleküls synthetisieren mussten, bei denen jeweils nur ein einzelnes Atom ausgetauscht wurde. Für deren Synthesen waren oft zehn und mehr Schritte der einzelnen Nukleosidbausteine notwendig. Erst dann konnte überprüft werden, welches der veränderten Ribozyme noch funktionsfähig ist. Daraus ließ sich auf die für die Reaktionskatalyse verantwortlichen Positionen in der RNA-Struktur schließen.
Links
- Atom-specific mutagenesis reveals structural and catalytic roles for active site-adenosine and hydrated Mg2+ in pistol ribozyme. Sandro Neuner, Christoph Falschlunger, Elisabeth Fuchs, Maximilian Himmelstoss, Aiming Ren, Dinshaw Patel, Ronald Micura. Angewandte Chemie International Edition 2017
- Structure-based insights into self-cleavage by a four-way junctional twister-sister ribozyme. Luqian Zheng, Elisabeth Mairhofer, Marianna Teplova, Ye Zhang, Jinbiao Ma, Dinshaw J. Patel, Ronald Micura, and Aiming Ren. Nature Communications 8, 1180 (2017)
- Micura Group
- Institut für Organische Chemie
- Forschungsschwerpunkt Centrum für Molekulare Biowissenschaften (CMBI)