Mit seinen Messmethoden dringt der Innsbrucker Chemiker Christoph Kreutz tief in die molekularen Strukturen des Lebens vor. Es gelingt ihm dabei das eigentlich Unsichtbare sichtbar zu machen. Mit Hilfe der Kernresonanz (NMR)-Spektroskopie können Strukturen die nur für sehr kurze Zeit - einige Millisekunden - und nur zu einem geringen Prozentsatz vorkommen in den Fokus gerückt werden. Denn während die Grundstrukturen der Nukleinsäuren sehr gut bekannt sind, sind Strukturveränderungen durch kleine chemische Veränderungen, wie sie etwa durch Umwelteinflüsse ausgelöst werden können, nicht immer leicht nachvollziehbar. „Durch diese kleine zusätzliche Gruppe verändert sich das Verhalten von Desoxyribonukleinsäure (DNA) auf eine völlig andere Weise als von Ribonukleinsäure (RNA). DNA verbleibt in einer Struktur ähnlich ihrer Grundstruktur, während RNA große strukturelle Veränderungen durchläuft. Wir untersuchen dabei Zustände, die die Nukleinsäuren nur sehr kurze Zeit, Milli- oder sogar nur Microsekunden, einnehmen“, erklärt Christoph Kreutz. „Ein Molekül kann sich zum Beispiel in zwei unterschiedliche Konformationen falten. Die Art der Faltung entscheidet dann auch über dessen Funktion und kann zum Beispiel den Informationsfluss in der Zelle beeinflussen.“ Deshalb ist es für die Wissenschaft wichtig, diese Zustände exakt zu beschreiben. Für die NMR-Messungen im Labor werden die gewünschten Moleküle synthetisch hergestellt und mit den notwendigen Isotopen markiert. „Die hochaufgelösten Strukturen von selten auftretenden Zuständen könnten uns neue Einsichten in die Funktion der RNA bei Prozessen wie der Ligandbindung oder der Katalyse ermöglichen“, verweist Christoph Kreutz auf die Vorteile der Methode, in der organische Chemie und Kernmagnetresonanz-Spektroskopie auf einzigartige Weise kombiniert werden.
Neue Einblicke
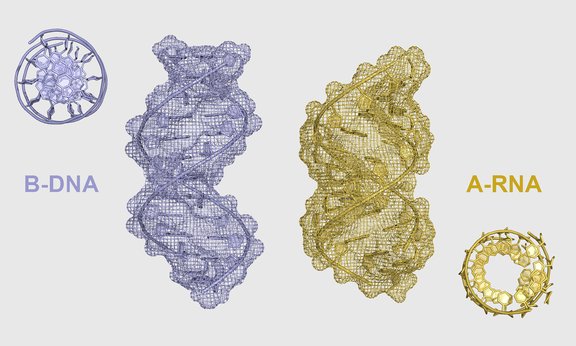
Die NMR-Methoden sind für Proteine schon länger etabliert, die Innsbrucker Forscher übertragen sie nun gemeinsam mit der Arbeitsgruppe um Hashim Al-Hashimi von der Duke University auf Nukleinsäuren. So lassen sich Strukturen von hochenergetischen Konformationen von RNA und DNA mit atomarer Auflösung beobachten. In einer aktuellen Arbeit in der Fachzeitschrift Nature Structural & Molecular Biology untersuchten die Forscher die beiden Nukleinsäure-Klassen und vergleichen den Einfluss von Methylierungen auf deren Struktur. Dabei zeigt sich, dass die minimale durch die chemische Veränderung ausgelöste Strukturänderung der DNA als Signal für Reparaturenzyme dient und damit sehr wichtig für den Erhalt der Erbgutinformation ist. „Durch die kurzfristige Strukturänderung bleibt die Stabilität der DNA letztlich erhalten“, erläutert Christoph Kreutz. Anders verhält es sich bei der kleinen Schwester der DNA, der RNA. Diese fungiert in der Zelle vor allem als Informationsüberträger. Die Messungen der Chemiker zeigen, dass die chemisch verursachten Veränderungen der RNA zu dauerhaften Strukturänderungen führen und somit die Funktion dieser Biomoleküle verändern. Das unterschiedliche Verhalten von DNA und RNA könnte auch erklären, warum die Natur die genetischen Informationen in der Doppelhelix der DNA abspeichert und nicht in der evolutionär viel älteren RNA. „Die DNA erweist sich als wesentlich flexibler und garantiert so die Stabilität des Erbguts“, sagt Christoph Kreutz. Sein Kooperationspartner Hashim Al-Hashimi ergänzt: „In diesen einfachen und schönen Strukturen ist eine verblüffende Komplexität eingebaut. Hier eröffnen sich ganz neue Dimensionen, für die wir bisher blind waren, weil uns die Werkzeuge gefehlt haben, um sie zu sehen.“
Die aktuellen Arbeiten entstanden in Kooperation mit Arbeitsgruppen an der Duke University, den Max F. Perutz Laboratories in Wien sowie dem University College London und wurden vom österreichischen Wissenschaftsfonds FWF finanziell gefördert.
Links
- Excited States of Nucleic Acids Probed by Proton Relaxation Dispersion NMR Spectroscopy. Michael Andreas Juen, Christoph Hermann Wunderlich, Felix Nußbaumer, Martin Tollinger, Georg Kontaxis, Robert Konrat, D. Flemming Hansen, Christoph Kreutz. Angew. Chem. Int. Ed. 2016, 55, 12008
- m1A and m1G disrupt A-RNA structure through the intrinsic instability of Hoogsteen base pairs. Huiqing Zhou, Isaac J Kimsey, Evgenia N Nikolova, Bharathwaj Sathyamoorthy, Gianmarc Grazioli, James McSally, Tianyu Bai, Christoph H Wunderlich, Christoph Kreutz, Ioan Andricioaei, and Hashim M Al-Hashimi. Nature Structural & Molecular Biology 23, 803–810 (2016)
- Kreutz Research Group
- Institut für Organische Chemie
- Forschungsschwerpunkt Centrum für Molekulare Biowissenschaften (CMBI)